I processen att genomföra genetiska studier stöter vi ofta på otillräckliga RNA-prover, till exempel för att studera små anatomiska orala tumörer, till och med encellsprover och prover av specifika genmutationer som transkriberas vid mycket låga nivåer i mänskliga celler.Naturligtvis, för covid-19-testet, om provtagningarna inte är på rätt plats eller inte tillräckligt många gånger under provtagningen, kommer provstorleken att vara mycket låg, vilket är anledningen till att kommissionen för hälsa och familjeplanering kom ut för två dagar sedan och klarade testet och om nukleinsyraprovtagaren inte tog sex prover kan du rapportera det.
Reagensens känslighet är viktig eftersom vi har det här problemet eller det problemet, så vad kan vi göra för att förbättra känsligheten för RT-PCR?
Innan vi diskuterar möjliga lösningar, låt oss nämna två stora komplikationer med den situation vi just nämnde.
Först och främst oroar vi oss för RNA-förlust när vi bara har ett fåtal cellpopulationer i vårt prov.Om traditionella separerings- och rengöringsmetoder används, såsom kolonnmetod eller nukleinsyrautfällningsmetod, är det stor risk att de få proverna går förlorade.En lösning är att lägga till en bärarmolekyl, såsom tRNA, men även då finns det ingen garanti för att vårt återhämtningsexperiment är OK.
Så vad är ett bättre sätt?Ett bra alternativ för odlade celler eller mikroanatomiska prover är att använda direkt lysering.
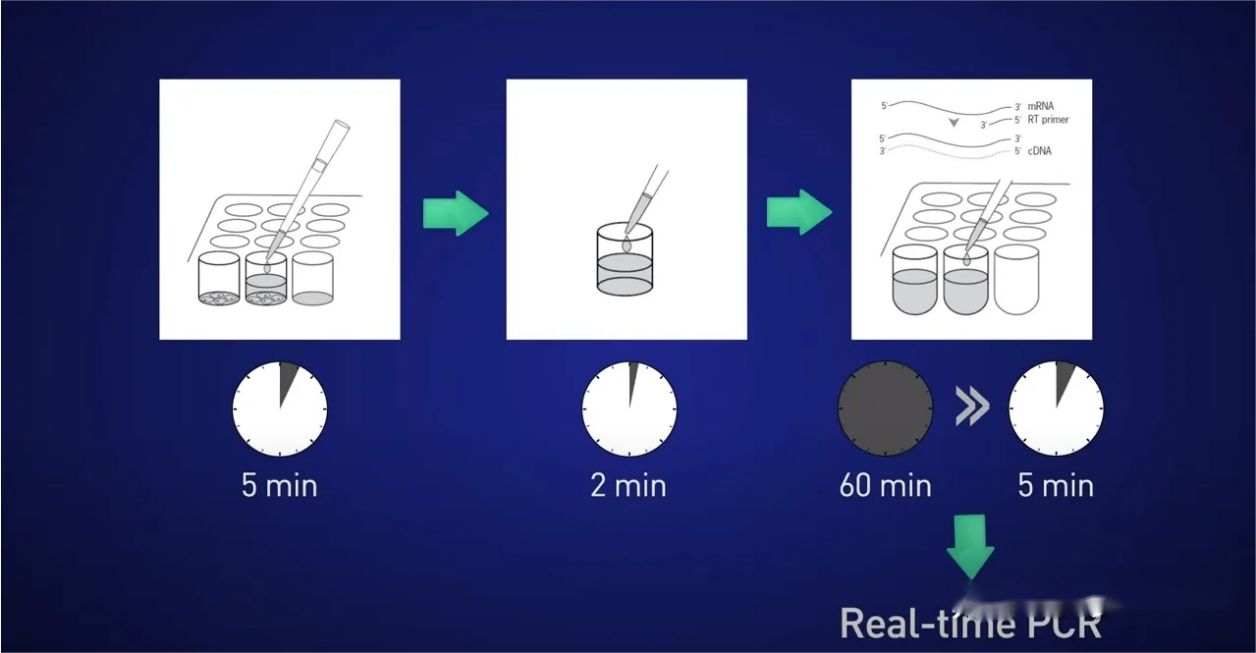
Tanken är att dela cellerna i 5 minuter, släppa RNA i lösningen, stoppa reaktionen i 2 minuter, sedan lägga till lysatet direkt till den omvända transkriptionsreaktionen så att inget RNA går förlorat, och slutligen sätta det resulterande cDNA direkt in i realtidsreaktionen.
Men vad händer om vi, på grund av en begränsad utgångspunkt eller en liten mängd målgenuttryck, kan återvinna allt RNA och ändå inte tillhandahålla tillräckligt med mallar för att få en bra realtidssignal?
I det här fallet kan förförstärkningssteget vara mycket användbart.
Följande är ett schema för att öka känsligheten efter omvänd transkription.Innan vi börjar måste vi fråga nedströms vilka mål vi är intresserade av, för att designa specifika primrar för dessa mål för pre-amplifiering.
Detta kan uppnås genom att skapa en blandad primer med upp till 100 par primers och en reaktionscykel på 10 till 14 gånger.Därför behövs en Master Mix speciellt utformad för detta krav för att förförstärka det erhållna cDNA:t.
Anledningen till att sätta antalet cykler mellan 10 och 14 är att detta begränsade antal cykler säkerställer slumpmässighet mellan de olika målen, vilket är avgörande för forskare som behöver kvantitativ molekylär information.
Efter pre-amplifiering kan vi få en stor mängd cDNA, så att detektionskänsligheten i back-end är avsevärt förbättrad, och vi kan till och med späda ut provet och utföra flera realtids-PCR-reaktioner för att eliminera möjliga slumpmässiga fel.
Posttid: 2023-apr-11